Updated April 2023. Created by Dr. Sergio Guajardo.
1. Recolección de muestras de suelo.
1.1. Materiales.
- Bolsas para muestras estériles de 1L
- Pala
- Pala portátil o de jardineria
- Etanol 70%
- Toalla de papel
- Estacas
- Cordín
- Regla de acero
- pHmetro para suelo (HI981030, HANNA)
- Medido de Conductividad para suelo (HI98331, HANNA)
- Guantes de latex
1.2. Procedimiento
Determinar un sitio de muestreo que contenga un numero suficiente de plantas (al menos 3 por especie) y establecer un punto central en el área de muestreo clavando una estaca para demarcarlo. Amarrar un cordín de tres metros a la estaca y tomar muestras dentro de este radio en todas las direcciones de forma aleatoria. Se considerarán como distintos sitios de muestreo, aquello lugares cuyos centros estén separados por 80-100 metros de distancia.
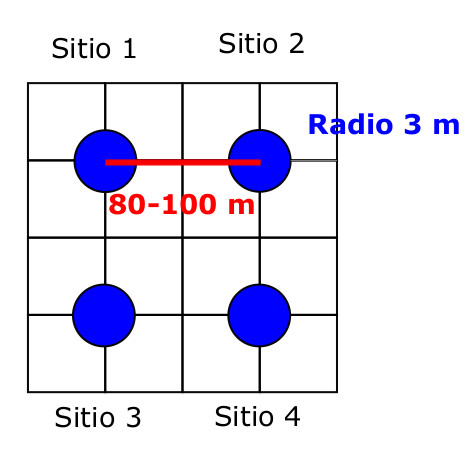
Para cada sitio de muestreo tomar el mismo numero de tipo de muestras distintas, por ejemplo, si son dos especies vegetales se recolectarán 3 rizósferas de la especie A, 3 rizósferas de la especie B y 3 muestras de suelo descubierto libre de plantas.
¡Utilizar guantes de latex!
Con la pala remover la zona superficial a muestrear, eliminado guijarros y material vegetal muerto. Medir el pH y la conductividad del suelo con el pHmetro (HI981030) y el medidor de conductividad (HI98331) respetivamente. Para esto hacer un agujero de 20 centímetros con la regla de acero y llenar con agua bidestilada estéril, insertar el pHmetro y luego el medidor de conductividad en el agujero con agua, medir y registrar.
Enterrar la pala en el suelo en un ángulo de 90 grados, sacar la pala verticalmente y nuevamente enterrar la pala en 90 grados ~5 cm atrás del primer agujero. Inclinar la pala hacia atrás para extraer un “core” de suelo. Con un cuchillo limpio (tanol al 70% y toalla de papel) seleccionar el suelo que esta entre 5 y 15 cm y guardar en una bolsa de muestra estéril.

En el caso del suelo rizosférico, remover la planta completa haciendo cortes con la pala en 90 grados por alrededor de la planta. Si es posible, remover la parte aérea de planta en terreno con un cuchillo y guardar las raíces con el suelo asociado en una bolsa estéril.
Limpiar la pala y el cuchillo entre cada muestra con etanol al 70% y toalla de papel.
2. Extracción de ácidos nucleicos para metagenomas celulares.
2.1 Materiales.
- Pinzas
- Espátula
- Tijera quirúrgica
- Tamiz Cisa de 4.75 mm (#4) y de 2 mm (#10).
- Colador de células de 100 μm estéril (EZFlow® Cell Strainer) o gasa estéril.
- Placas de Petri desechables
- Tubos cónicos de 50mL estériles
- Buffer PBS 1X
- Criotubos 1.5 mL
2.2 Procedimiento.
Todo este procedimiento debe realizarse en campana de flujo laminar en condiciones de esterilidad.
Para extraer ácidos nucleicos es necesario separar el suelo rizosférico del suelo que no esta unido fuertemente a las raíces. Para esto, tomar la planta con una pinza y sacudir fuertemente dentro de la bolsa de muestreo en una campana de flujo laminar. El suelo que quedo unido a las raíces será considerado suelo rizosférico y el que se soltó dentro de la bolsa se denominará RSS.
Las raíces que contienen el suelo rizósferico serán separadas y depositadas en un tubo cónico de 50 mL que contiene 15 mL de buffer PBS 1X usando pinza y tijera. Mezclar por vortex suave 2 minutos y trasvasar la suspensión de rizósfera a un tubo nuevo con un colador de células o gasa esteril. Agregar 15 mL de buffer PBS 1X al tubo que contiene las raíces y mezclar por vortex 2 minutos, trasvasando el contenido al mismo tubo del paso anterior con un colador de células o gasa estéril (repetir 2 veces).
Centrifugar el tubo con la rizósfera resuspendida por 10 minutos a 4000g en centrifuga refrigerada a 4 ºC. Eliminar el sobrenadante y guardar el tubo con el pellet a -20 ºC.
Tomar 0.5 mL de pellet con una espátula estéril depositándolo en un criotubo, repetir el proceso con 0.5 mL de pellet adicionales para obtener un total de ~1 gramo de rizósfera.
Para el RSS y el suelo descubierto mezclar cada bolsa de muestra por agitación y cribar una parte del RSS y suelo utilizando un tamiz de malla de acero de 4.75 y 2 mm. Recuperar el suelo en una bolsa de muestra esteril abierta (abrir la bolsa a su maxima extención con tijera) y transferir a un tubo conico de 50 mL y guardar a -20 ºC.
Con una espátula esteril tomar 0.5 mL de suelo depositándolo en un criotubo, repetir con 0.5 mL adicionales para obtener un total de ~ 1 gramo de RSS o suelo.
Todo el material de metal incluyendo tamiz, espatulas, pinzas y tijeras deben ser esterilizadas entre cada muestra utlizando etanol 70% y flameando el material en la llama del mechero.
2.3 Extracción de ADN
Para extraer ADN utilizaremos el kit DNeasy PowerSoil Pro Kit (47014, Qiagen) utilizando 1 gramo de suelo, RSS o rizósfera (para suelos más ricos en materia orgánica se puede partir de 250 mg y utilizar una sola columna).
La extracción se realiza siguiendo las instrucciones del fabricante excepto porque se harán dos extracciones de 500 mg cada una y se juntarán en una sola columna para su elución.
El ADN extraído se cuantificará utilizando un fluorímetro Qubit y el kit para cuantificación de ADN doble hebra de alta sensibilidad (Q32854, Invitrogen). La concentración mínima necesaria para secuenciar por shotgun son 10-100 ng totales de ADN.
3. Extracción de ácidos nucleicos para metagenomas virales.
3.1 Materiales.
Pinzas
Espátula
Tijera quirúrgica
Tamiz de 5 mm de acero (Tamices Cisa #4).
Placas de Petri desechables
Tubos cónicos de 50mL estériles
Tampón AKC’ (Citrato de potasio 1% P/V, PBS 1X 10% V/V, Sulfato de Magnesio 150 mM, pH 7)
Filtros Sterivex 0.2 µm PES (SVGP01050, Merck)
Jeringas esteriles de 50 mL
DNAsa I (A3778, ITW Reagents)
EDTA 1M
Filtro 100 kDa (UFC810024, Merck o VS04T4, Sartorius)
PEG8000 (V3011, Promega)
NaCl
DNeasy PowerSoil Pro Kit (47014, Qiagen)
Micropipeta P1000
Micropipeta P200
Puntas estériles con filtro de 200 µL
Puntas estériles con filtro de 1000 µL
Solución PBS-BSA 1% (37525, Thermo)
3.2 Procedimiento.
Para las extracciones de ADN viral se necesita un mínimo de 10 gramos de suelo rizosférico o suelo descubierto por lo que para el suelo rizosférico no es posible separar la rizósfera y el RSS.
Para el suelo rizosferico tomar la planta con una pinza y cortar las raíces con el suelo adherido sin sacudir. Cribar por malla de acero de 5 mm, recuperar el suelo rizosférico tamizado en placa de Petri.
Para el suelo descubierto, mezclar agitando la bolsa y cribar por malla de acero de 5 mm, recuperar el suelo tamizado en placa de Petri.
Resuspender 10 gramos de suelo rizosférico o suelo en 15 mL de tampón AKC’ y mezclar por inversión durante 30 segundos para incubar a 4 ºC por toda la noche.
Agitar por vortex a velocidad media 1 minuto, luego agitar de forma horizontal en agitador orbital a 400 rpm por 30 minutos.
Centrifugar a 3000g por 10 minutos en centrifuga refrigerada a 4 ºC y recuperar el sobrenadante en un tubo nuevo.
Volver a resuspender el pellet de suelo rizosférico o suelo en 10 mL de tampón AKC’ y en agitar en el agitador orbital a 400 rpm por 30 minutos.
Centrifugar a 3000g por 10 minutos en centrifuga refrigerada a 4 ºC y recuperar el sobrenadante en un tubo nuevo.
Volver a resuspender el pellet en 10 mL de tampón AKC’ y repetir los pasos de agitación y centrifugado.
Al final de este proceso se debería tener un total de 30 mL de sobrenadante por cada 10 gramos de suelo rizosférico o suelo descubierto.
Filtrar los 30 mL de sobrenandante por un filtro Sterivex de 0.2 µm PES usando una jeringa de 50 mL.
Tratar con DNAsa I 3000U por 10 gramos de suelo durante toda la noche a 4 ºC, inactivar la DNAsa con EDTA a una concentración final de 100mM.
Concentrar la muestra utilizando filtración tangencial en filtros de 100 kDa o por precipitación con PEG8000, dependiendo del tipo de muestra.
Si el método elegido es filtración tangencial, el filtro debe ser preparado antes de su utilización, para esto llenar con agua bidestilada estéril y centrifugar a 3000g. Eliminar cualquier remanente de agua e incubar el filtro con 2 mL de PBS-BSA 1% por al menos 4 horas a 4 ºC. Eliminar el PBS-BSA y lavar el filtro 3 veces con agua bidestilada estéril dejando el filtro listo para ser usado. Trasvasar 4 mL de la muestra por vez al filtro tangencial y centrifugar por 2 minutos a 1500g. Rellenar el filtro con muestra y centrifugar por 2 minutos a 1500g, repetir hasta que en el filtro solo queden 500 µL de muestra. Cada vez que se rellena el filtro es necesario balancear con respecto al contrapeso o el resto de los tubos. Si luego de 2 minutos de centrifugación el volumen no ha disminuido, se puede aumentar la velocidad de centrifugación a 3000g y el tiempo de centrifugación a lo que sea necesario.
Sacar el filtro del tubo colector y cubrir el filtro exterior con Parafilm. Agitar por vortex a 1500 rpm durante 1 minuto, y recuperar el volumen con una micropipeta P200 en un criotubo de 1.5 mL resuspendiendo suavemente. Agregar 500 µL de el liquido filtrado (el que quedo en el tubo colector) y repetir la agitación por vortex, recuperando el volumen con una micropipeta P200 en el mismo criotubo. A final de este paso se debe tener un criotubo con 1 mL de muestra.
En el caso de utilizar PEG8000, añadir solucion de PEG8000-NaCl (*20% PEG8000, NaCl 2,5 M) al sobrenadante en una razón de 1:1 (1 volumen de PEG8000-NaCl por 1 volumen de sobrenadante filtrado) e incubar a 4 ºC durante toda la noche. Centrifugar las muestras a 10000g durante 30 min (o 5000g por 1 hora) y eliminar el sobrenadante con micropipeta (ser cuidadoso porque el pellet puede ser no visible). Resuspender el pellet con 750 µL de tampón PowerBeads del kit DNeasy PowerSoil Pro (47014, Qiagen).
*Preparar la solución disolviendo primero el NaCl y luego el PEG8000 en agitador magnético)
3.3 Extracción de ADN.
Para extraer ADN utilizaremos el kit DNeasy PowerSoil Pro Kit (47014, Qiagen) utilizando como volumen de partida 2x500 µL de la muestra concentrada en el paso anterior (filtración tangencial). El material de partida es liquido por lo que al usar un volumen mayor a 250 µL es posible que las distintas soluciones del kit se diluyan con respecto a su concentración optima. La extracción se realiza siguiendo las instrucciones del fabricante excepto si se usa mas de 500 µL de muestra inicial. En este caso se hará dos extracciones de 500 µL y se juntaran en una sola columna para su elución.
Si la concentración de partículas virales se hizo con PEG8000, trasvasar el pellet resuspendido en el tampón de PowerBeads al tubo de PowerBeads para continuar la extracción siguiendo las instrucciones del fabricante.
El ADN extraído se cuantificará utilizando un fluorimetro Qubit y el kit para cuantificación de ADN doble hebra de alta sensibilidad (Q32854, Invitrogen). La concentración mínima necesaria para secuenciar por shotgun son 10-100 ng totales de ADN.
