Updated April 2023. Created by Dr. Sergio Guajardo.
1. Recolección de muestras de aire.
1.1. Materiales.
- Coriolis µ
- Base para muestreo prolongado
- Trípode
- Fuente de energía (corriente o generador Honda EU10i)
- Botella de inyección con filtro de 0.2 µm
- Mangueras de inyección
- Cono de recolección estéril (autoclavado) o lavado (Agua bidestilada estéril apirógena, Hipoclorito de sodio 1%, etanol 70%)
- Tampón de recolección estéril (Glicerol 2% autoclavado) idealmente diluir el glicerol en agua bidestilada estéril apirógena o agua miliQ.
- Tubos cónicos de 15 mL estériles.
- Criotubos de 1.5 mL
- Micropipeta P1000
- Puntas P100 estériles con filtro
- RNA later
- Agua bidestilada estéril apirógena o agua miliQ
- Hipoclorito de sodio 1%
- Etanol 70%
- Toalla de papel
- Espátula delgada
1.2 Procedimiento.
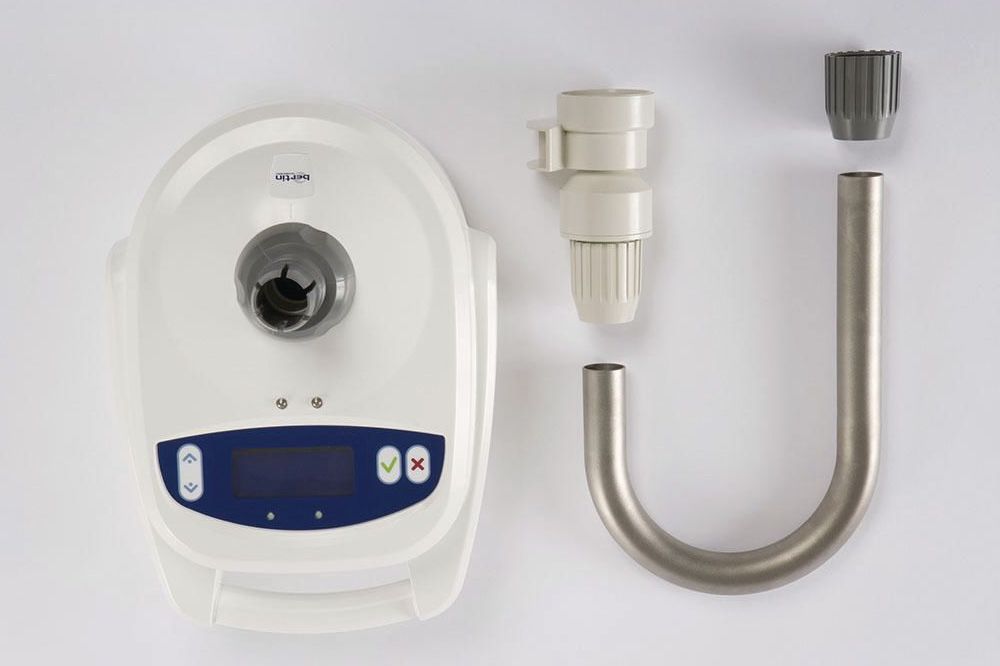
Ensamblar el equipo Coriolis µ (incluyendo el cono de recolección) siguiendo las instrucciones descritas en el manual del fabricante y montar sobre base de alimentación para muestreo prolongado (instalada previamente en trípode). Revisar que la entrada de la boquilla de recolección quede en orientación contraria al asa del equipo.

Encender el equipo manteniendo presionado el botón con el símbolo “√” de color verde hasta que aparezca el dialogo de autotest. Una ves concluidos los autotest, el equipo esta listo para funcionar. Llenar el cono de colección usando la función priming del equipo, a la que se accede presionando la flecha que apunta hacia arriba “^”. Para llenar el cono de colección mantener presionado el botón con el símbolo “√” de color verde hasta completar 7.5 mL (según graduación del cono). Para salir del modo priming se debe presionar el botón de flecha hacia abajo “v”.
Una vez devuelta a la pantalla principal establecer el tiempo de recolección (240 min), velocidad de recolección (300L/min) y tiempo de espera (0 min). Luego comenzar recolección de la muestra donde aparecerá la opción de seleccionar el volumen de inyección de tampón por minuto.
El volumen de tampón a inyectar depende de dos variables principales, la temperatura y humedad relativa, por lo que es recomendable probar el volumen que se inyectara. Para determinar el volumen correcto se debe muestrear sin inyectar tampón, registrando el volumen inicial y el final luego de 10 minutos de muestreo, dividiendo el volumen perdido por 10 se obtendrá el volumen de inyección. Si no es posible determinar el volumen de inyección, un buen punto de partida son 0.5 mL/min. Revisar cada 0.5-1 hora de muestreo para ajustar volumen de inyección (pausando el equipo con el botón “√” de color verde) y evitar que el volumen de tampón en el cono sobrepase los 10 mL durante el periodo de muestreo y 7,5 mL al final del muestreo.
Cuando el tiempo de muestreo haya concluido (240 min) el equipo se detendrá solo, luego de esto revisar el informe de corrida y apagar presionando el botón “X” de color rojo.
Es importante que el volumen de recolección final sea 7,5 mL (si el volumen es menor, se puede usar la función priming para completar). Agregar RNA later a una concentración final de 25% v/v (2,5 mL de RNA later por 7,5 mL de muestra) utilizando micropipeta P1000 y puntas con filtro estériles. Trasvasar a un tubo de 15 mL estéril y guardar a 4ºC por máximo una semana. Para periodos de almacenamiento mayores se recomienda guardar a -20 ºC.
1.2 Limpieza del equipo.
Una vez terminada la recolección es necesario limpiar algunas partes del equipo, para esto se usará hipoclorito de sodio al 1% (solución debe estar fresca, menos de 3 días), agua bidestilada apirógena y etanol 70%. Se limpiará la cañería metálica, la boquilla de colección, las mangueras de inyección, la botella de tampón y los conos de recolección. Limpiar primero con hipoclorito de sodio al 1%, poner especial cuidado con la boquilla que usualmente junta polvo y suciedad. De ser necesario se puede eliminar la suciedad con una espátula envuelta en toalla de papel remojada en hipoclorito de sodio. Luego enjuagar con agua bidestilada apirógena y por último lavar con etanol al 70% dejando secar al aire. Adicionalment, en caso de estar en laboratorio, las piezas se autoclavan envueltas en papel kraft y se secan en horno de secado.
Para “limpiar” el interior del equipo, se debe cargar el cono con 15 mL de etanol 70% y dejar el equipo muestreando (sin inyección de volumen) hasta que el cono quede seco (siempre queda un remanente). Luego de esto apagar el equipo y limpiarlo exteriormente con toalla de papel y etanol 70%.
2. Extracción de ácidos nucleicos.
2.1. Materiales.
- Filtro 100 kDa (UFC810024, Merck o VS04T4, Sartorius)
- Criotubo de 1.5 mL
- Solución PBS-BSA 1% (37525, Thermo)
- Vortex de 1500 rpm (88880017, Thermo)
- Parafilm
- Micropipeta P200
- Micropipeta P1000
- Puntas estériles con filtro de 200 µL
- Puntas estériles con filtro de 1000 µL
- DNeasy PowerSoil Pro Kit (47014, Qiagen)
- Trizol LS (10296, Thermo)
- Cloroformo (102445, Merck)
- Etanol (100983, Merck)
- E.Z.N.A.® MicroElute RNA Clean-up Kit (R6247, Omega Bio-Tek)
- TURBO DNA-free™ Kit (AM1907, Invitrogen)
- Qubit dsDNA HS Assay Kit (Q32854, Invitrogen)
- Qubit RNA HS Assay Kit (Q32852, Invitrogen)
2.2 Concentración de la muestra.
Para extraer ácidos nucleicos es necesario concentrar las muestras, por lo que se utilizará filtración tangencial usando un dispositivo de 100 kDa. El filtro tangencial debe ser preparado antes de su utilización, para esto llenar con agua bidestilada estéril y centrifugar a 3000g. Eliminar cualquier remanente de agua e incubar el filtro con 2 mL de PBS-BSA 1% por al menos 4 horas a 4 ºC. Eliminar el PBS-BSA y lavar el filtro 3 veces con agua bidestilada estéril dejando el filtro listo para ser usado. Trasvasar 4 mL de la muestra al filtro tangencial y centrifugar por 2 minutos a 1500g. Rellenar el filtro con muestra y centrifugar por 2 minutos a 1500g, repetir hasta que en el filtro solo queden 500 µL de muestra. Cada vez que se rellena el filtro es necesario balancear con respecto al contrapeso o el resto de los tubos. Sacar el filtro del tubo colector y cubrir el filtro exterior con Parafilm. Agitar por vortex a 1500 rpm durante 1 minuto, y recuperar el volumen con una micropipeta P200 en un criotubo de 1.5 mL resuspendiendo suavemente. Agregar 500 µL de el liquido filtrado (el que quedo en el tubo colector) y repetir la agitación por vortex, recuperando el volumen con una micropipeta P200 en el mismo criotubo. A final de este paso se debe tener un criotubo con 1 mL de muestra.
2.3 Extracción de ADN.
Para extraer ADN utilizaremos el kit DNeasy PowerSoil Pro Kit (47014, Qiagen) utilizando como volumen de partida 250 a 500 µL de la muestra concentrada en el paso anterior. El material de partida es liquido por lo que al usar un volumen mayor a 250 µL es posible que las distintas soluciones del kit se diluyan con respecto a su concentración optima. La extracción se realiza siguiendo las instrucciones del fabricante excepto si se usa mas de 500 µL de muestra inicial. En este caso se hará dos extracciones de 250 µL y se juntaran en una sola columna para su elución.
El ADN extraído se cuantificará utilizando un fluorimetro Qubit y el kit para cuantificación de ADN doble hebra de alta sensibilidad (Q32854, Invitrogen). La concentración mínima necesaria para secuenciar por shotgun son 10-100 ng totales de ADN.
2.4 Extracción de ARN.
Para extraer ARN se debe utilizar el total de la muestra (1mL) para esto se debe dividir la muestra en 4 alícuotas de 250µL en tubos de 1.5 mL.
A cada tubo con 250 µL de muestra agregar 750 µL de Trizol LS frio y resuspender por pipeteo suave.
Incubar por 5 minutos a temperatura ambiente para completar la lisis y agregar 200 µL de cloroformo mezclando el tubo por inversión.
Incubar por 3 minutos y centrifugar 15 minutos a 12.000g en centrifuga refrigerada a 4 ºC.
Recuperar la fase acuosa (superior) y proseguir con la limpieza del ARN usando el kit E.Z.N.A (R6247, Omega Bio-Tek).
Añadir 1 volumen de etanol absoluto (100983, Merck) por cada volumen de muestra (250 µL de etanol por 250µL de muestra) y mezclar por inversión.
Pasar la muestra ya mezclada con etanol (la capacidad máxima de la columna es de 700 μL, se pueden cargar volúmenes mayores sucesivamente) por la columna del kit E.Z.N.A (R6247, Omega Bio-Tek) siguiendo las instrucciones del fabricante “Centrifugar a 10.000g durante 15 segundos a temperatura ambiente”. Repetir este paso hasta que toda la muestra haya sido pasada por la columna y seguir con el protocolo “Desalting Protocol” del fabricante en el paso 7.
Una vez eluido el ARN, esté se tratará con TURBO DNase (AM1907, Invitrogen) para eliminar el ADN remanente siguiendo las instrucciones del fabricante.
El ARN extraído se cuantificará utilizando un fluorímetro Qubit y el kit para cuantificación de ARN de alta sensibilidad (Q32852, Invitrogen). La concentración mínima necesaria para secuenciar por shotgun son 500 ng totales de ARN.
